L’imagerie médicale : un bref aperçu de son rôle et de ses limites
Aujourd’hui en France, près de 64 millions d’examens d’imageries sont prescrits par an. Il s’agit majoritairement de la radiographie et de l’échographie, mais aussi des examens d’IRM, CT-scan, PET-scan, etc. L’analyse de ces images médicales permet la détection et la segmentation des régions d’intérêts afin d’identifier et localiser avec précision les structures anatomiques, les tumeurs ou les lésions, ainsi que la reconnaissance des pathologies. Ainsi, l’imagerie médicale est aujourd’hui une composante indispensable de la médecine moderne, permettant de visualiser les structures internes du corps, de détecter des anomalies et d’orienter le diagnostic et le traitement.
Par leur capacité à identifier des structures et des relations complexes dans ces données, les algorithmes d’apprentissage profond (Deep Learning) ont bouleversé les approches traditionnelles. Ces algorithmes atteignent des performances remarquables dans des tâches de segmentation, de détection et de classification, couvrant une large gamme de modalités d’imagerie et de cas cliniques. Ainsi, en 2024, 81% des radiologues déclarent utiliser l’IA (l’intelligence artificielle), majoritairement pour faire de l’aide au diagnostic1.
Cependant, les performances de ces modèles sont fortement conditionnées par la disponibilité de jeux de données d’entraînement de haute qualité, annotés par des experts. En effet, la généralisation des modèles à différentes populations de patients reste un défi majeur, et dépend de la représentativité, de la diversité et de la quantité des images utilisées durant l’entraînement. Or, dans le domaine médical, la constitution de tels jeux de données s’avère complexe. D’une part, les contraintes éthiques liées à l’anonymisation des données de santé et au respect de la RGPD (règlement général de protection des données) complexifient l’accès aux données. À noter que la proposition d’un nouvel espace européen des données de santé, approuvée en début d’année, vise à faciliter l’accès aux données médicales, notamment dans un but de recherche et d’innovation. Cette proposition est accompagnée d’un renforcement des règles d’anonymisation et de la sécurité pour protéger les données personnelles. D’autre part, la labellisation (annotation précise de chaque image) et le contrôle de sa qualité nécessite un investissement important en temps et en ressources humaines, par la mobilisation d’experts médicaux. On observe également un fort déséquilibre des bases de données, où les pathologies rares sont fréquemment sous-représentées, ce qui accentuent encore la problématique de généralisation du modèle.
Pour surmonter ces limitations, la génération et l’utilisation des données synthétiques comme complément des données réelles est explorée. En permettant de créer des ensembles de données variés et à grande échelle, ces approches ouvrent la voie à une meilleure représentativité des jeux de données et à une amélioration des performances des modèles d’apprentissage profond.
La génération de données synthétiques
Il existe diverses méthodes pour générer des données synthétiques2, parmi lesquelles :
- L’utilisation de modèles génératifs, tels que les GANs (Generative Adversarial Networks), les VAEs (Variational Autoencoders) et les modèles de diffusion, représente la méthode la plus commune. Ces techniques, bien que puissantes, nécessitent des ressources de calcul importantes et restent fortement dépendantes de la qualité des jeux de données d’entraînement, tout comme leurs homologues dédiés au diagnostic médical. Les GANs, en particulier, sont sujets à des phénomènes d’« effondrement de mode », où le générateur produit des images très similaires entre elles, limitant ainsi la diversité du jeu de données synthétique. Ils peuvent également introduire des hallucinations (productions d’éléments incohérents ou irréalistes dans une image synthétique), limitant la vraisemblance des images produites.
- La génération de nouvelles images en combinant des pixels ou des segments provenant d’images différentes. On peut citer par exemple la technique du mixup, qui génère une combinaison pondérée de paires d’images. Cette méthode s’est révélée efficace pour améliorer la généralisation des modèles.
- Les méthodes de génération basées sur la reconstruction intervenant directement sur les données brutes, avant leur reconstruction (par exemple dans le cas des scanners 3D). Elles permettent de simuler des artefacts (par exemple de mouvement) ou de reconstruire des images 2D à partir de volumes 3D (par exemple, des radiographies à partir de CT-scans). Cependant, ces techniques nécessitent un accès aux données brutes avant reconstruction, qui ne sont généralement pas conservées en milieu clinique.
- Les techniques basées sur des modèles (model-based methods) représentent une approche plus flexible. Elles reposent sur la modélisation des patients, permettant de contrôler l’anatomie et la physiologie humaines désirées, ainsi que sur l’utilisation de simulateurs d’imagerie reproduisant les principes physiques et les processus des scanners médicaux pour générer des images réalistes. Il est ensuite possible d’injecter une lésion artificielle dans un modèle de patient afin de simuler une pathologie spécifique. En intégrant directement des contraintes anatomiques dans les images générées, ces techniques permettent de produire des variations aléatoires représentatives de la variabilité inter-sujets, ou encore de simuler des artefacts courants d’acquisition, renforçant ainsi la robustesse des algorithmes d’analyse d’images lors de l’entrainement.
Focus sur l’approche par simulateur physique
Afin d’illustrer le potentiel de cette dernière technique, prenons l’exemple d’un projet de détection de nodules pulmonaires. Les nodules pulmonaires sont une lésion du poumon constitués d’un tissu pathologique de forme généralement arrondie inférieure à 3 cm. Ces nodules sont généralement bénins, mais demandent un suivi, et peuvent nécessiter une intervention chirurgicale si l’un d’eux se révèle être malin. Ces nodules sont habituellement détectés à la suite d’un examen radiologique. Le taux de détection de ces nodules par un radiologiste est d’environ 64%3.
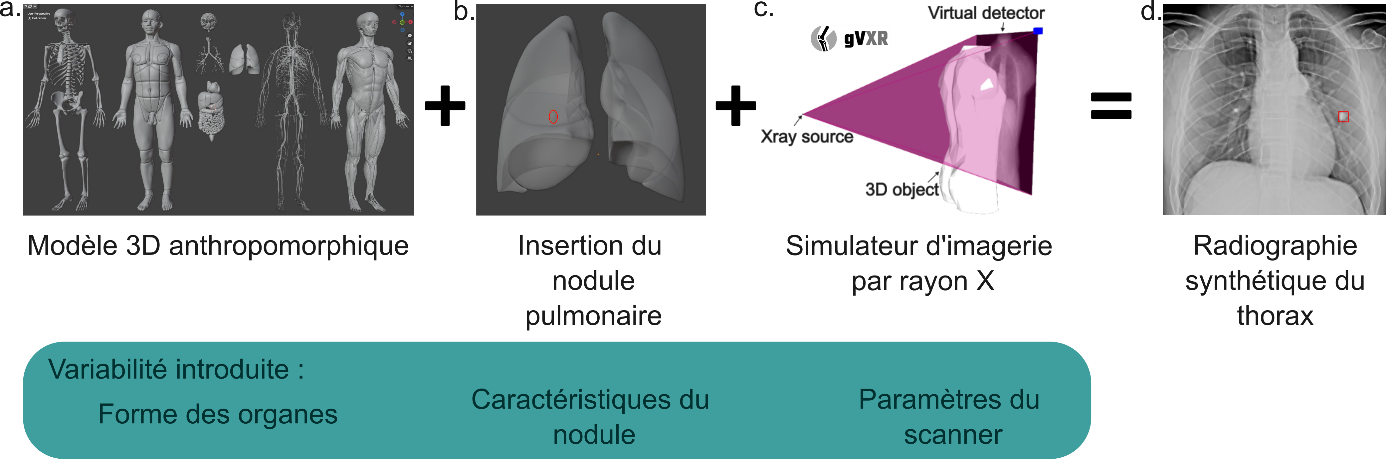
La simulation de radiographies est possible grâce à des simulateurs avancés, ici gVirtualXRay4, qui repose sur la loi de Beer-Lambert pour modéliser l’absorption des rayons X par les tissus traversés. En combinant cet outil à des modèles anthropomorphiques virtuels, il est possible de générer rapidement de larges ensembles d’images synthétiques labellisées, ouvrant la voie à l’entraînement de modèles d’apprentissage profond sur des jeux de données de tailles plus importantes.
Des radiographies synthétiques sont ensuite générées à l’aide du modèle Z-Anatomy5, issu de la segmentation de l’IRM d’un homme adulte. Ce modèle reproduit fidèlement les structures anatomiques clés (poumons, cœur, trachée, etc.). Des variations aléatoires de la taille des organes et de la densité des tissus sont introduites pour simuler la diversité anatomique des patients. Des nodules pulmonaires, représentés par des sphères de tailles et de densités variées, sont ajoutés aléatoirement au niveau du poumon, imitant ainsi des pathologies réelles.
L’algorithme d’apprentissage profond choisi ici est YOLOv8m6 (détection et classification d’objets). Les paramètres d’évaluation de l’efficacité du modèle sont la sensibilité (% de nodules détectés) et la précision (% de détections correspondant bien à un nodule). Dans des travaux récents, l’enrichissement d’un jeu de données réelles par des données synthétiques a permis d’améliorer de façon significative les performances de l’algorithme et d’atteindre des performances supérieures à celles d’un radiologue :
- Après un entraînement sur un jeu de données composé de 100% de radiographies réelles, le modèle atteint une sensibilité de 55,7 % et une précision de 68,5 %.
- Après un entrainement sur un jeu de données composé de 20% de données réelles, et 80% de données synthétiques, la sensibilité atteint 73,8 % et la précision 77,6 %.
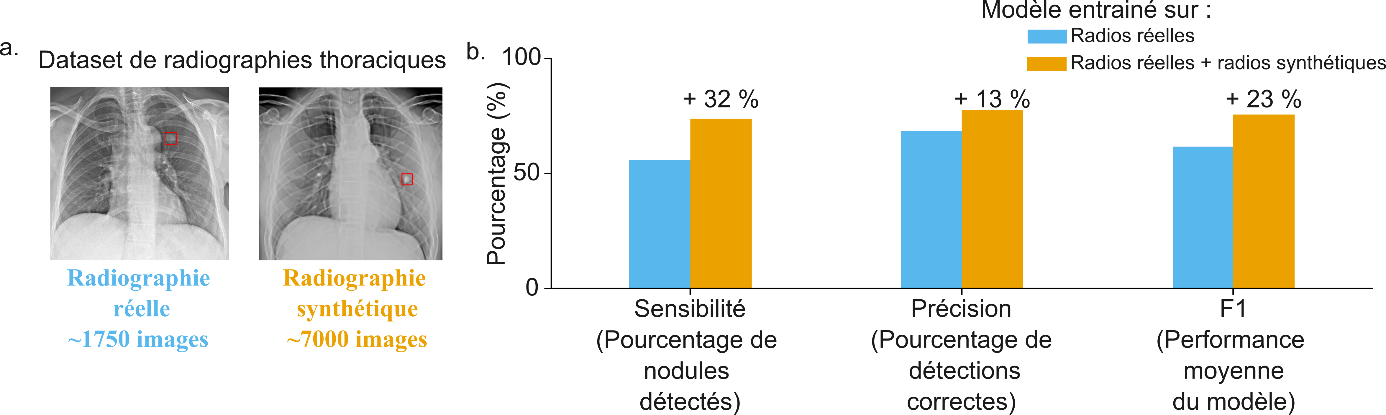
Ces résultats montrent le potentiel des données synthétiques pour renforcer les performances des algorithmes d’apprentissage profond. Cette approche peut facilement être étendue à d’autres pathologies, ouvrant la voie à des outils de diagnostic plus performants et accessibles.
Des perspectives prometteuses
La génération d’images synthétiques pour l’imagerie médicale est un domaine en plein essor qui commence à se démocratiser, avec l’émergence de jeux de données synthétiques en libre accès7-9. L’utilité de ces images dépasse largement l’amélioration des performances des modèles de diagnostic, en particulier pour les méthodes dites « model-based ». Ces méthodes présentent en effet l’avantage de permettre une conversion facile d’images d’une modalité d’imagerie à une autre, en modifiant le simulateur d’imagerie utilisé. Cette flexibilité ouvre la voie à une intégration de différents examens issus de modalités d’imagerie différentes par les modèles de diagnostiques.
Les images synthétiques générées peuvent aussi être utilisées pour évaluer et affiner les technologies d’imagerie « hors-ligne », améliorant leur précision et leur efficacité pour mieux répondre aux besoins cliniques.
Ces images synthétiques peuvent également être employées à des fins pédagogiques, pour la formation des étudiants en médecine.